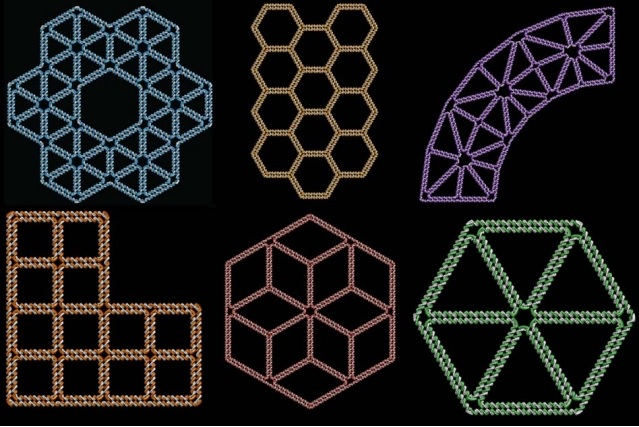
Investigadores del MIT y la Universidad Estatal de Arizona han diseñado un programa de computadora que permite a los usuarios traducir cualquier dibujo de forma libre en una estructura bidimensional a nanoescala hecha de ADN.
Hasta ahora, el diseño de tales estructuras ha requerido experiencia técnica que pone el proceso fuera del alcance de la mayoría de las personas.
Usando el nuevo programa, cualquiera puede crear una nanoestructura de ADN de cualquier forma, para aplicaciones en biología celular, fotónica, detección cuántica y computación, entre muchos otras.
“Lo que este trabajo hace es permitir que cualquiera pueda dibujar literalmente cualquier forma 2-D y convertirla en origami de ADN automáticamente”, dice Mark Bathe, profesor asociado de ingeniería biológica en el MIT y autor principal del estudio.
Los investigadores publicaron sus hallazgos en Science Advances, y el programa, llamado PERDIX, está disponible en línea.
Los autores principales del artículo son Hyungmin Jun, postdoctoral del MIT, y Fei Zhang, profesora asistente de investigación en la Universidad Estatal de Arizona. Otros autores son el investigador asociado del MIT, Tyson Shepherd, el reciente receptor de doctorado del MIT, Sakul Ratanalert, el científico investigador asistente de la ASU Xiaodong Qi y el profesor de la ASU, Hao Yan.
El origami de ADN, la ciencia de plegar el ADN en estructuras diminutas, se originó a principios de la década de 1980, cuando Ned Seeman, de la Universidad de Nueva York, propuso aprovechar las capacidades de apareamiento de bases del ADN para crear arreglos moleculares arbitrarios.
En 2006, Paul Rothemund de Caltech creó las primeras estructuras de ADN bidimensionales con andamios, tejiendo una larga hebra única de ADN (el andamio) de forma tal que las cadenas de ADN conocidas como “grapas” se hibridarán para ayudar a la estructura a mantener su forma.
Otros utilizaron más tarde un enfoque similar para crear complejas estructuras tridimensionales de ADN.
Sin embargo, todos estos esfuerzos requerían un diseño manual complicado para encaminar el andamio a través de toda la estructura y generar las secuencias de las hebras cortadas.
En 2016, Bathe y sus colegas desarrollaron una manera de automatizar el proceso de generación de una estructura de ADN poliédrica en 3D y, en este nuevo estudio, se propusieron automatizar el diseño de estructuras de ADN en 2D de forma arbitraria.
Para lograr eso, desarrollaron un nuevo enfoque matemático para el proceso de enrutar el andamio de una sola hebra a través de toda la estructura para formar la forma correcta.
El programa de computadora resultante puede tomar cualquier dibujo de forma libre y traducirlo en la secuencia de ADN para crear esa forma y en las secuencias para las hebras de la grapa.
La forma se puede esbozar en cualquier programa de dibujo por computadora y luego convertirla en un archivo de diseño asistido por computadora (CAD), que se alimenta al programa de diseño de ADN.
“Una vez que tiene ese archivo, todo es automático, muy parecido a la impresión, pero aquí la tinta es ADN”, dice Bathe.
Una vez que se generan las secuencias, el usuario puede ordenar que fabriquen fácilmente la forma especificada.
En este artículo, los investigadores crearon formas en las que todos los bordes constan de dos dúplex de ADN, pero también tienen un programa de trabajo que puede utilizar seis dúplex por borde, que son más rígidos.
La herramienta de software correspondiente para los poliedros tridimensionales, llamada TALOS, está disponible en línea y se publicará próximamente en la revista ACS Nano.
Las formas, que varían de 10 a 100 nanómetros de tamaño, pueden permanecer estables durante semanas o meses, suspendidas en una solución tampón.
“El hecho de que podamos diseñar y fabricar estos de una manera muy simple ayuda a resolver un importante cuello de botella en nuestro campo”, dice Bathe.
“Ahora, el campo puede hacer la transición hacia grupos mucho más amplios de personas en la industria y el mundo académico, pudiendo funcionalizar estructuras de ADN y desplegarlas para diversas aplicaciones”.
Debido a que los investigadores tienen un control tan preciso sobre la estructura de las partículas de ADN sintético, pueden unir una variedad de otras moléculas en ubicaciones específicas.
Esto podría ser útil para templar antígenos en patrones a nanoescala para arrojar luz sobre cómo las células inmunitarias reconocen y se activan mediante disposiciones específicas de antígenos que se encuentran en virus y bacterias.
“La forma en que las células inmunes reconocen los patrones a nanoescala de los antígenos es un área de la inmunología poco conocida”, dice Bathe.
“Agregar antígenos a las superficies de estrucuturas ADN para mostrarlas en patrones organizados es una forma poderosa de probar esa biología”.
Otra aplicación clave es diseñar circuitos de captación de luz que imiten los complejos fotosintéticos que se encuentran en las plantas.
Para lograrlo, los investigadores están uniendo tintes sensibles a la luz conocidos como cromóforos a los andamios de ADN.
Además de la recolección de luz, dichos circuitos también podrían utilizarse para realizar detección cuántica y cálculos rudimentarios.
Si tiene éxito, estos serían los primeros circuitos de computación cuántica que pueden operar a temperatura ambiente, dice Bathe.
Otras aplicaciones posibles para las estructuras de ADN incluyen su uso para ayudar a organizar los ensamblajes de proteínas macromoleculares que se encuentran en las células, para que se puedan obtener imágenes más fácilmente con la microscopía crioelectrónica de alta resolución.
Fuente: MIT News