La tecnología de edición es precisa y ampliamente aplicable a todos los tejidos y especies animales.
Investigadores de la Universidad de Duke han desarrollado una herramienta de edición basada en ARN que se dirige a células individuales, en lugar de genes.
Es capaz de apuntar con precisión a cualquier tipo de célula y agregar selectivamente cualquier proteína de interés.
Los investigadores dijeron que la herramienta podría permitir modificar células y funciones celulares muy específicas para controlar enfermedades.
Usando una sonda basada en ARN, un equipo dirigido por el neurobiólogo Z. Josh Huang, Ph.D. y el investigador postdoctoral Yongjun Qian, Ph.D. demostraron que pueden introducir etiquetas fluorescentes en las células para marcar tipos específicos de tejido cerebral.
Un interruptor de encendido/apagado sensible a la luz para silenciar o activar las neuronas de su elección; e incluso una enzima de autodestrucción para eliminar con precisión algunas células pero no otras.
Su sistema de control y seguimiento celular selectivo se basa en la enzima ADAR, que se encuentra en las células de todos los animales.
Si bien estos son los primeros días de CellREADR (Acceso celular a través de la detección de ARN por ADAR endógeno), las posibles aplicaciones parecen ser infinitas, dijo Huang, al igual que su potencial para trabajar en todo el reino animal.
“Estamos entusiasmados porque esto proporciona una tecnología simplificada, escalable y generalizable para monitorear y manipular todos los tipos de células en cualquier animal”, dijo Huang.
“De hecho, podríamos modificar tipos específicos de función celular para controlar enfermedades, independientemente de su predisposición genética inicial”, dijo.
“Eso no es posible con las terapias o medicamentos actuales”.
CellREADR es una cadena personalizable de ARN compuesta por tres secciones principales: un sensor, una señal de alto y un conjunto de planos.
En primer lugar, el equipo de investigación decide qué tipo de célula específica quiere investigar e identifica un ARN objetivo que es producido únicamente por ese tipo de célula.
La notable especificidad tisular de la herramienta se basa en el hecho de que cada tipo de célula fabrica ARN característico que no se ve en otros tipos de células.
Luego se diseña una secuencia sensora como la hebra complementaria del ARN objetivo.
Así como los peldaños del ADN están formados por moléculas complementarias que se atraen de forma inherente entre sí, el ARN tiene el mismo potencial magnético para unirse con otra pieza de ARN si tiene moléculas coincidentes.
Después de que un sensor ingresa a una célula y encuentra su secuencia de ARN objetivo, ambas piezas se unen para crear una pieza de ARN de doble cadena.
Este nuevo mashup de ARN activa la enzima ADAR para inspeccionar la nueva creación y luego cambiar un solo nucleótido de su código.
La enzima ADAR es un mecanismo de defensa celular diseñado para editar el ARN de doble cadena cuando ocurre, y se cree que se encuentra en todas las células animales.
Sabiendo esto, Qian diseñó la señal de alto de CellREADR utilizando las mismas ediciones específicas de ADAR de nucleótidos en el ARN de doble cadena.
La señal de alto, que impide que se construyan los planos de proteínas, solo se elimina una vez que el sensor de CellREADR se acopla a su secuencia de ARN objetivo, lo que lo hace altamente específico para un tipo de célula determinado.
Una vez que ADAR elimina la señal de alto, los planos pueden ser leídos por la maquinaria celular que construye la nueva proteína dentro de la célula objetivo.
En su artículo, Huang y su equipo pusieron a CellREADR a prueba.
“Recuerdo hace dos años cuando Yongjun construyó la primera iteración de CellREADR y la probó en el cerebro de un ratón”, dijo Huang.
“Para mi asombro, funcionó espectacularmente en su primer intento”.
La planificación y el diseño cuidadosos del equipo valieron la pena, ya que luego pudieron demostrar que CellREADR etiquetó con precisión poblaciones específicas de células cerebrales en ratones vivos, así como monitores de actividad agregados e interruptores de control donde se indicaba.
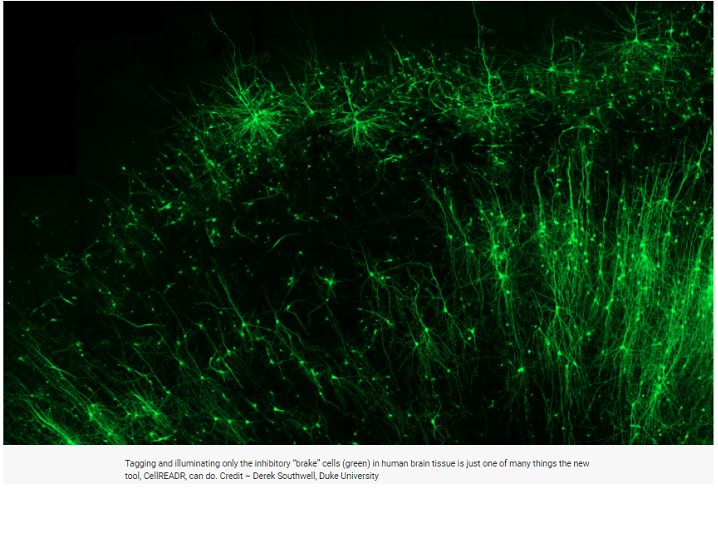
También funcionó bien en ratas y en tejido cerebral humano recolectado de cirugías de epilepsia.
“Con CellREADR, podemos elegir poblaciones para estudiar y realmente comenzar a investigar la gama completa de tipos de células presentes en el cerebro humano“, dijo el coautor Derek Southwell, M.D., Ph.D., neurocirujano y profesor asistente en el departamento de neurocirugía de Duke.
Southwell espera que CellREADR mejore su comprensión y la de otros sobre el diagrama de cableado de los circuitos del cerebro humano y las células dentro de ellos y, al hacerlo, ayude a promover nuevas terapias para los trastornos neurológicos, como un método nuevo y prometedor para tratar la epilepsia resistente a los medicamentos.
Huang y Qian están especialmente esperanzados con el potencial de CellREADR como un “medicamento de ARN programable” para posiblemente curar enfermedades, ya que eso es lo que los atrajo a ambos a la ciencia en primer lugar.
Han solicitado una patente sobre la tecnología.
“Cuando me especialicé en farmacología como estudiante universitario, era muy ingenuo”, dijo Qian.
“Pensé que podías hacer muchas cosas, como curar el cáncer, pero en realidad es muy difícil. Sin embargo, ahora pienso, sí, tal vez podamos hacerlo”.
Fuente: Duke Today